Die komplizierten molekularen Prozesse, die den zellulären Veränderungen während der pflanzlichen Immunität zugrunde liegen, weisen ein hohes Maß an proteomischer Plastizität auf und beinhalten die De-novo-Synthese von regulatorischen Proteinen sowie deren regulierten Abbau. Daher ist es unumgänglich, das enge Zusammenspiel zwischen Proteinsynthese, -transport und -abbau zu untersuchen und zu erforschen, wie diese Wege ihre Funktionen zur Regulierung der Pflanzenimmunität koordinieren. In diesem Zusammenhang haben wir entdeckt, dass der regulierte Proteinumsatz über das Ubiquitin-Proteasom-System (UPS) mehrere Aspekte der Pflanzenimmunität steuert (Üstün et al., 2016). Eine bedeutende Rolle des UPS bei der Regulierung der Pflanzenimmunität wird auch durch die Erkenntnisse gestützt, dass Pflanzenpathogene Strategien entwickelt haben, um das UPS für ihre Virulenz zu nutzen. Es wurde gezeigt, dass mehrere bakterielle Typ-III-Effektorproteine (T3Es) die pflanzliche Abwehr unterdrücken, indem sie als E3-Ligasen fungieren (z. B. AvrPtoB) oder die Ubiquitinierung von Zielproteinen fördern (z. B. HopM1). Ein direkterer Weg zur Umgehung der UPS wird durch bakterielle Typ-III-Effektoren beschritten, die direkt mit Proteasom-Komponenten interagieren und dadurch die Proteasom-Funktion unterdrücken, um die Salicylsäure (SA)-vermittelte Pflanzenimmunität zu unterdrücken (Üstün et al., 2013; Üstün et al., 2014; Üstün & Börnke, 2015). Kürzlich haben wir gezeigt, dass Pst das Proteasom durch ein bestimmtes Effektor-Repertoire hemmt, darunter HopM1, das mit UPS-Komponenten zu assoziieren scheint (Üstün et al., 2016).
Neben dem UPS ist die Autophagie ein weiterer wichtiger Abbauweg, der vor allem bei der zellulären Homöostase, der Stresstoleranz und der Pathogenabwehr in eukaryontischen Organismen eine Rolle spielt. Autophagie-Mechanismen stützen sich auf einen Kernsatz konservierter AuTophaGy-verwandter (ATG) Gene zur Bildung von Doppelmembranvesikeln, die als Autophagosomen bezeichnet werden und zytoplasmatische Ladung absondern und an lytische Kompartimente (d. h. Lysosomen/Vakuolen) zum Abbau und Recycling weiterleiten (Üstün et al., 2017). Inzwischen gibt es immer mehr Hinweise darauf, dass die Autophagie auch als selektiver Mechanismus zum Abbau von Proteinaggregaten, Organellen und spezifischen Proteinen dient, z. B. bei der zellulären Qualitätskontrolle und unter Stressbedingungen. Die Selektivität wird durch Autophagie-Adaptoren wie NBR1 (Neighbour of BRAC1) vermittelt, der an ubiquitylierte Substrate und Autophagosom-assoziierte ATG8-Proteine bindet. Jüngste Erkenntnisse haben gezeigt, dass das Proteasom durch die Autophagie (Proteaphagie") als Reaktion auf chemische Proteasom-Inhibition und Nährstoffmangel eliminiert wird, was auf eine funktionelle Verbindung zwischen den beiden proteolytischen Systemen in Pflanzen hindeutet. Kürzlich haben wir festgestellt, dass das pflanzenpathogene Bakterium Pseudomonas syringae pv. tomato DC3000 (Pst) die Autophagie aktiviert und zum Abbau des Proteasoms durch den Proteaphagie-Weg führt, um seine Pathogenität zu erhöhen (Üstün et al., 2018).
Auf der Grundlage unserer Ergebnisse legen wir nahe, dass Pflanzenpathogene auf die beiden wichtigsten proteolytischen Abbauwege abzielen, um weitreichende Auswirkungen auf die Pflanze zu haben und möglicherweise das Dreieck Proteinsynthese-Transport-Abbau zu beeinflussen. Die vom Krankheitserreger ausgelöste Aktivierung der Autophagie und die Unterdrückung der Proteasomfunktion führen zu einem massiven Abbau und/oder einer Anhäufung von Targets, die an der Verteidigung und anderen zellulären Prozessen beteiligt sind. Weitere Auswirkungen der Autophagie-Aktivierung und Proteasom-Suppression auf Prozesse wie die Proteinsekretion und -translation sind derzeit noch unbekannt und werden im Üstün-Labor untersucht. Ein weiteres Ziel des Üstün-Labors ist es, zu verstehen, wie die Wirkung von T3Es proteolytische Wege modulieren kann. Auf der Grundlage vorläufiger und früherer Ergebnisse werden wir Effektorproteine als Werkzeuge einsetzen, um den Autophagie-Weg zu entschlüsseln und neue Regulatoren der Autophagie zu entdecken.
Die proteomische Analyse möglicher Ziele der pathogen-induzierten Autophagie führte zu der Entdeckung, dass neben dem Proteasom auch andere Signalwege wie der RNA-Stoffwechsel, die Protein-Translation und der Vesikel-Transport Ziele oder Regulatoren der Autophagie sein könnten. Auf der Grundlage dieser vorläufigen Daten haben wir eine Komponente des Vesikeltransports als einen neuen Modulator der Autophagie identifiziert (Gouguet et al., unveröffentlicht). Es hat den Anschein, dass der Verlust dieser und anderer Trafficking-Komponenten und die Störung der Proteinsekretion an den ER-Ausgangsstellen die Autophagie-Reaktionen moduliert, was eine neue Ebene des Zusammenspiels zwischen dem Endomembransystem und dem Autophagie-Mechanismus offenbart.
Unter Verwendung von Pseudomonas- und Xanthomonas-Effektoren haben wir diese systematisch auf ihre Fähigkeit untersucht, die Proteasomfunktion und die Autophagie zu beeinträchtigen. IP-MS-Experimente mit Effektor-Kandidatenproteinen ergaben eine Untergruppe von Effektoren, die mit Autophagie-Komponenten interagieren. Bisher haben wir einen Xanthomonas-Effektor identifiziert, der in der Lage ist, eine Autophagie-Komponente anzugreifen, um den Autophagie-Umsatz zu modulieren (Leong et al., in Vorbereitung), den wir mit zellbiologischen und biochemischen Ansätzen weiter charakterisieren werden. Wir werden unsere Studien auf andere Effektorproteine ausdehnen und setzen derzeit die CRISPR/Cas9-Technologie ein, um Tomatenpflanzen mit veränderten proteolytischen Profilen zu erzeugen.
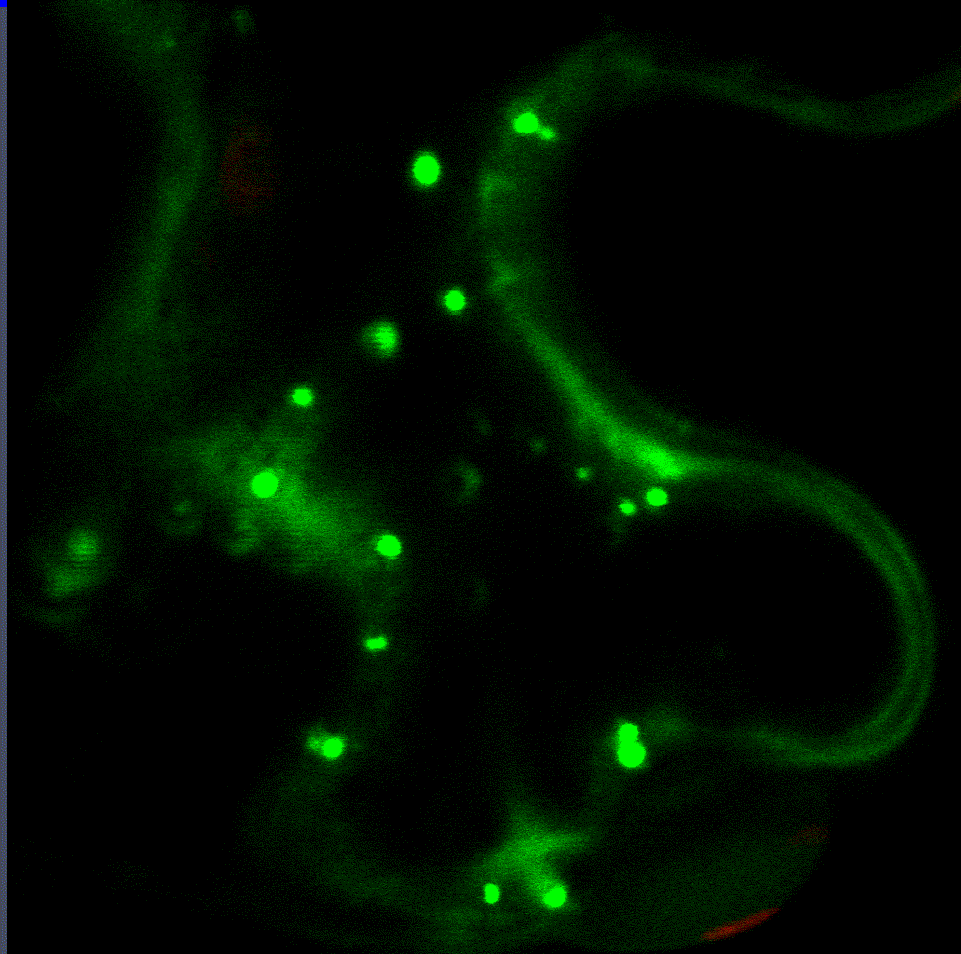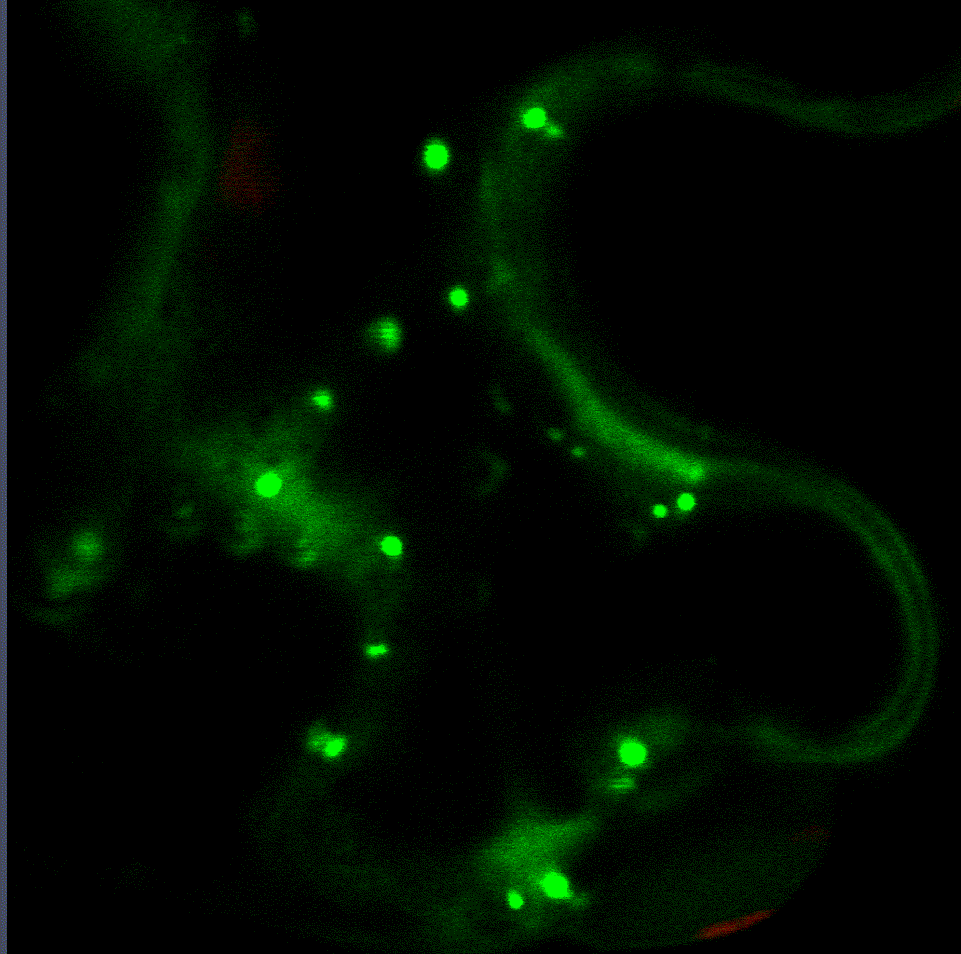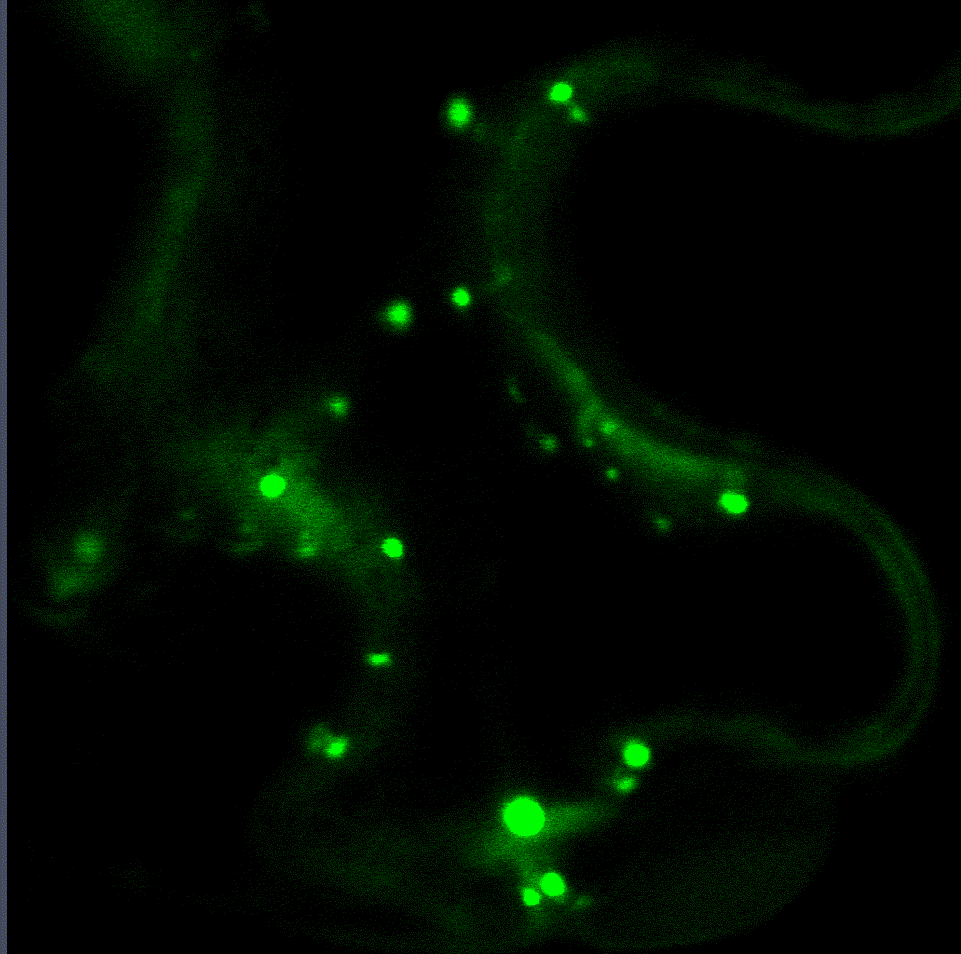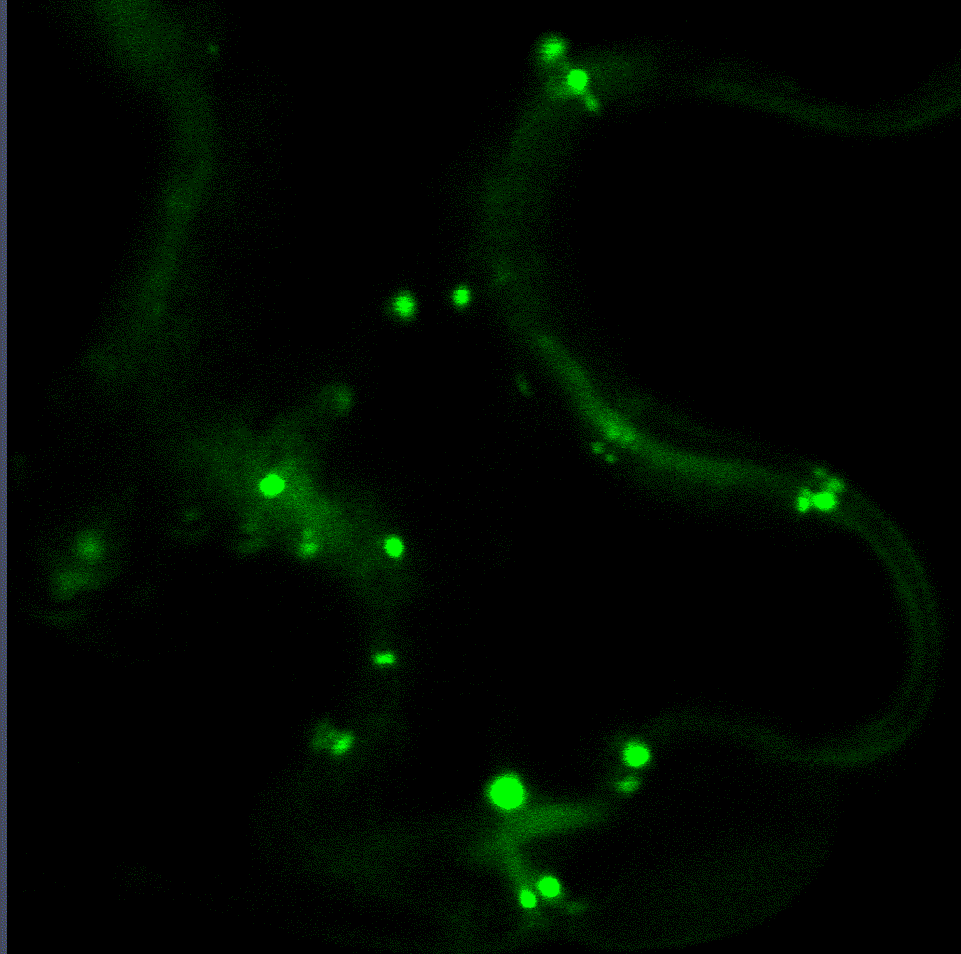
Die regulatorische Feinabstimmung des Proteasoms ist von zentraler Bedeutung für das Ergebnis von Interaktionen zwischen Pflanzen und Mikroben und scheint eine empfindliche Kombination aus Down- und Up-Regulierung seiner Aktivität zu sein. Daher analysieren wir derzeit in einem vom CRC1101 finanzierten Projekt die mechanistischen Erkenntnisse darüber, wie das Proteasom während der pflanzlichen Immunität auf transkriptioneller und posttranslationaler Ebene reguliert wird. Im Rahmen dieses Projekts haben wir zwei Transkriptionsfaktoren identifiziert, die die Proteasom-Genexpression während einer bakteriellen Infektion regulieren (Langin et al., unveröffentlicht). Interaktionsstudien mit beiden Transkriptionsfaktoren ergaben, dass verschiedene Proteine an ihrem Abbau über das ERAD-System und am Transport vom ER in den Zellkern beteiligt sind. Derzeit untersuchen wir weitere Zielgene dieser Transkriptionsfaktoren, da sie offenbar den Kompromiss zwischen Proteasom-Aktivierung, Wachstum und Verteidigung vermitteln.

Um sich vor diesen Krankheitserregern zu schützen, wird die pflanzliche Immunität effizient auf transkriptioneller und posttranskriptioneller Ebene reguliert. Die Kompartimentierung von mRNA in translatorisch unterdrückte Aggregate, so genannte Processing Bodies (PBs), ist ein wichtiger posttranskriptioneller Regulierungsprozess, der an der Entwicklung und an Stressreaktionen beteiligt ist. Vorläufige Ergebnisse haben gezeigt, dass das bakterielle Pflanzenpathogen Pseudomonas syringae (Pst) die Bildung von PB bei einer Infektion in einer effektorabhängigen Weise induziert und dass Arabidopsis-Pflanzen mit PB-Defekten toleranter gegenüber Pst sind. Dies deutet darauf hin, dass PBs negative Regulatoren der pflanzlichen Immunität sind, die von den bakteriellen Effektoren angegriffen werden können. Das vorgeschlagene Projekt zielt genau darauf ab, die Rolle der PBs als post-transkriptionelle Regulatoren der Pflanzenimmunität und die Fähigkeit der Pst-Effektoren, sie zu modulieren, zu untersuchen. Zu diesem Zweck wird eine Kombination aus genetischen, biochemischen, proteomischen und zellbiologischen Ansätzen durchgeführt, um Folgendes zu untersuchen: 1) die Dynamik und Rolle von PBs, 2) die durch Effektoren vermittelte Modulation der PB-Bildung und 3) das Zusammenspiel zwischen PBs und Autophagie, alles im Kontext einer kompatiblen Pflanze-Bakterium-Interaktion.
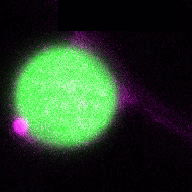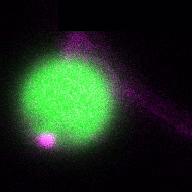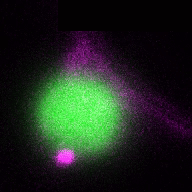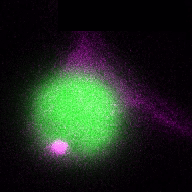
In einem umfassenderen Szenario sind Pflanzen ständig verschiedenen pathogenen und nützlichen Mikroben ausgesetzt, und daher ist es von entscheidender Bedeutung, das Bakteriom (Mikrobiom) in diese Gleichung einzubeziehen, um ein ganzheitliches Bild der Rolle der Autophagie in den Interaktionen zwischen Pflanzen und Mikroben zu erhalten. Das Bild wird noch komplexer, wenn wir die zelltypspezifische Autophagie-Reaktion auf der Pflanzenseite betrachten. Indem wir untersuchen, wie das Mikrobiom und die verschiedenen Zelltypen die Autophagie beeinflussen und umgekehrt, werden wir ein tieferes Verständnis der Rolle der Autophagie bei Interaktionen zwischen Pflanzen und Mikroben gewinnen. Daher werden wir im Rahmen des ERC Starting Grant geförderten Projekts DIVERSIPHAGY folgende Fragen und Ziele untersuchen:
In DIVERSIPHAGY werden wir eine Kombination aus modernsten biochemischen, proteomischen, einzelligen transkriptomischen und zelltypspezifischen reversen genetischen Ansätzen verwenden, um die Rolle der Autophagie in den Interaktionen zwischen Pflanzen und Mikroben zu entschlüsseln. Unser Ziel ist es, einen ganzheitlichen Überblick über die Rolle der Autophagie bei der Interaktion zwischen Pflanzen und Mikroben zu gewinnen, indem wir die bakterielle, genetische und zelluläre Vielfalt nutzen, wobei der Schwerpunkt auf zelltyp- und organspezifischen Autophagie-Reaktionen liegt.